Julia を導入
————————————
ベイズ推論による機械学習入門 須山 敦志 著 を読む
————————————
内容としては、機械学習とベイズ推論の基礎を復習するのに良かった。
あとは、本書籍のサポートサイト、著者のサポートサイトを参考に進めるが、サンプルプログラムはJuliaで記述されている。というわけで、Juliaで対応してみる。以前にも経験済だが、最新のver1.2では、色々と仕様が変更されており、書籍の執筆された時点での環境との不一致か、なかなか安定した動作が期待できないのと、貼付のDockerファイルもMacではハングして達成できない。仕方無しにJulia ver 0.7にダウングレードしてみた。
https://julialang.org/からMac OX S用をダウンロードすればよいが、現時点での最新版は1.2だが、ver 0.4, 0.7から相当に改変があり、ここでは、学習プログラムを動かすためにあえて、Older Releasesからver0.7をダウンロードしてインストールする。
(注:あとから気がついたが、ver 0.6.4にして、そのままコードをjupyterに落とせば、何の問題もなく作動!)
以下のパスを.bashrc に追記
|
1 |
export PATH=${PATH}:/Applications/Julia-0.7.app/Contents/Resources/julia/bin |
$ source .bashrc
以下のエリアスは、.bash_profileに追記
|
1 |
echo "alias julia='/Applications/Julia-0.7.app/Contents/Resources/julia/bin/julia' |
$ source .bash_profile
Jupyter Notebookでの使用は、Juliaを立ち上げ、
|
1 2 3 4 5 6 |
using Pkg Pkg.add("IJulia") Pkg.build("IJulia") using IJulia IJulia.notebook() |
|
1 2 3 4 5 6 7 8 |
Pkg.add("PyPlot") Pkg.build("PyPlot") Pkg.add("PyCall") Pkg.build("PyCall") Pkg.add(Distributions") Pkg.build("Distributions") |
|
1 2 |
using PyPlot, PyCall using Distributions |
|
1 2 3 4 5 6 7 8 9 10 |
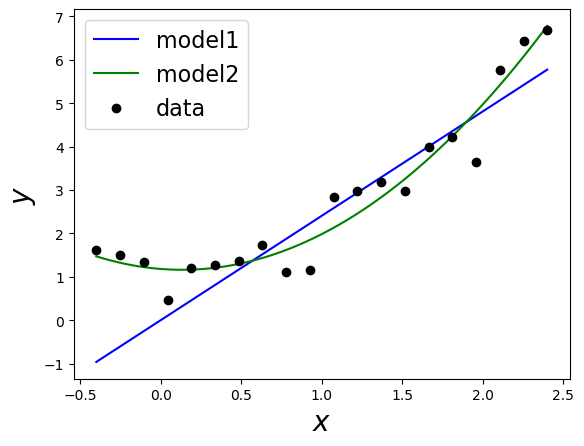
# true param W = Array([1.0, 0.0, 1.0]) # generate data sigma = 0.5 N = 20 X = linspace(-0.4,2.4,N) Y = [W[1] + W[2]*x + W[3]*x^2 + sigma*randn() for x in X] X_min = minimum(X) X_max = maximum(X) |
Out:2.4
|
1 2 3 4 |
# regression1 X_all = linspace(X_min, X_max, 100) W1 = sum(Y.*X) / sum(X.^2) Y1 = [W1*x for x in X_all] |
Out: 100-element Array{Float64,1}:
-0.9624213660385834
-0.8943713704600977
-0.826321374881612
-0.7582713793031263
-0.6902213837246407
-0.6221713881461549
-0.5541213925676692
-0.48607139698918356
-0.4180214014106979
-0.34997140583221215
-0.28192141025372647
-0.21387141467524076
-0.14582141909675506
?
5.025978244868158
5.094028240446643
5.162078236025129
5.230128231603614
5.298178227182101
5.366228222760586
5.434278218339072
5.502328213917558
5.570378209496043
5.638428205074529
5.706478200653015
5.7745281962315005
|
1 2 3 4 5 6 7 |
# regression2 X2 = zeros(3, N) X2[1,:] = 1 X2[2,:] = X X2[3,:] = X.^2 W2 = inv(X2*X2') * X2*Y Y2 = [W2[1] + W2[2]*x + W2[3]*x^2 for x in X_all] |
Out:100-element Array{Float64,1}:
1.4716160624130428
1.4395658262082924
1.4092644768125695
1.3807120142258738
1.3539084384482056
1.3288537494795643
1.3055479473199505
1.2839910319693637
1.2641830034278045
1.2461238616952723
1.2298136067717678
1.2152522386572902
1.2024397573518402
?
5.345933981351435
5.467785784341084
5.591386474139762
5.716736050747467
5.8438345141641985
5.972681864389958
6.103278101424744
6.235623225268559
6.3697172359213985
6.505560133383267
6.643151917654164
6.782492588734086
|
1 2 3 4 5 6 7 8 9 |
# show data figure() plot(X_all, Y1, "b-") plot(X_all, Y2, "g-") plot(X, Y, "ko") legend(["model1","model2","data"], loc="upper left", fontsize=16) xlabel("\$x\$", fontsize=20) ylabel("\$y\$", fontsize=20) show() |
次にパッケージLoisticRegressionを追加
Juliaを起動し、]を押してパッケージモードにしてから、
|
1 |
generate LoisticRegression |
LogisticRegressionフォルダのsrcの中のLogisticRegression.jlを、ダウンロード済のLogisticRegression.jlに入れ替える。
一度Juliaを終了し、LogisticRegressionディレクトリに入ります。
Juliaを起動して]を押してパッケージモードにして、パッケージの中で利用するパッケージ、ここではDistributionsを以下のようにしてManifest.tomlに登録。
|
1 2 |
activate . add Distributions |
delキーを押してパッケージモードを終了した後、
|
1 |
using LogisticRegression |
ジュピターを立ち上げて、環境OKなる。
|
1 |
notebook(detached=true) |
LogisticRegressionのファルダの置かれているPathに要注意して、
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 |
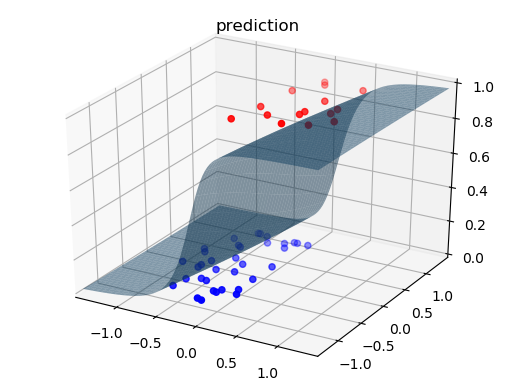
using PyPlot, PyCall using Distributions push!(LOAD_PATH, "/Users/*****/Desktop/BayesBook-master/src/LogisticRegression") import LogisticRegression <pre> function visualize_surface(mu, rho, X, Y, text) N = 100 R = 100 xmin = minimum(X[1,:]) xmax = maximum(X[1,:]) ymin = minimum(X[2,:]) ymax = maximum(X[2,:]) lx = xmax - xmin ly = ymax - ymin xmin = xmin - 0.25 * lx xmax = xmax + 0.25 * lx ymin = ymin - 0.25 * ly ymax = ymax + 0.25 * ly x1 = linspace(xmin,xmax,R) x2 = linspace(ymin,ymax,R) x1grid = repmat(x1, 1, R) x2grid = repmat(x2', R, 1) val = [x1grid[:] x2grid[:]]' z_list = [] sigma = log.(1 + exp.(rho)) for n in 1 : N W = rand(MvNormal(mu, diagm(sigma.^2))) z_tmp = [LogisticRegression.sigmoid(W'*val[:,i]) for i in 1 : size(val, 2)] push!(z_list, z_tmp) end z = mean(z_list) zgrid = reshape(z, R, R) # 3D plot figure("surface") clf() plot_surface(x1grid, x2grid, zgrid, alpha=0.5) scatter3D(X[1,Y.==1], X[2,Y.==1], Y[Y.==1]+0.01, c="r", depthshade=true) scatter3D(X[1,Y.==0], X[2,Y.==0], Y[Y.==0], c="b", depthshade=true) xlim([xmin, xmax]) ylim([ymin, ymax]) zlim([0, 1]) title(text) end |
Out:visualize_surface (generic function with 1 method)
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 |
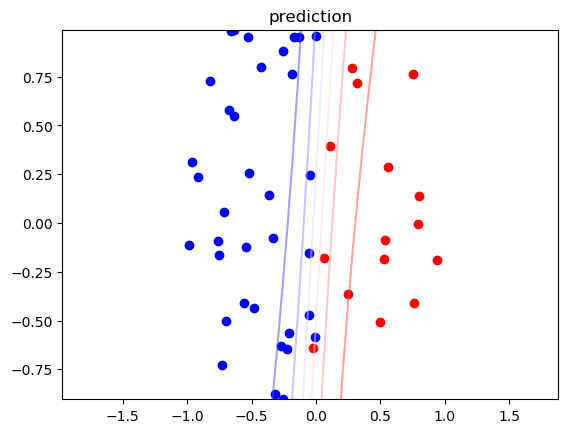
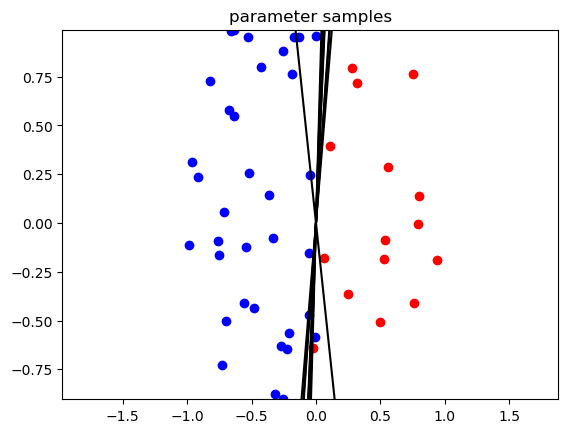
function visualize_contour(mu, rho, X, Y) N = 100 R = 100 xmin = 2*minimum(X[1,:]) xmax = 2*maximum(X[1,:]) ymin = minimum(X[2,:]) ymax = maximum(X[2,:]) x1 = linspace(xmin,xmax,R) x2 = linspace(ymin,ymax,R) x1grid = repmat(x1, 1, R) x2grid = repmat(x2', R, 1) val = [x1grid[:] x2grid[:]]' z_list = [] W_list = [] sigma = log.(1 + exp.(rho)) for n in 1 : N W = rand(MvNormal(mu, diagm(sigma.^2))) z_tmp = [LogisticRegression.sigmoid(W'*val[:,i]) for i in 1 : size(val, 2)] push!(W_list, W) push!(z_list, z_tmp) end z = mean(z_list) zgrid = reshape(z, R, R) # precition figure("contour") clf() contour(x1grid, x2grid, zgrid, alpha=0.5, cmap=get_cmap("bwr")) scatter(X[1,Y.==1], X[2,Y.==1], c="r") scatter(X[1,Y.==0], X[2,Y.==0], c="b") xlim([xmin, xmax]) ylim([ymin, ymax]) title("prediction") # parameter samples figure("samples") clf() for n in 1 : 10 draw_line(W_list[n], xmin, xmax) end scatter(X[1,Y.==1]', X[2,Y.==1]', c="r") scatter(X[1,Y.==0]', X[2,Y.==0]', c="b") xlim([xmin, xmax]) ylim([ymin, ymax]) title("parameter samples") end |
Out:visualize_contour (generic function with 1 method)
|
1 2 3 4 5 |
function draw_line(W, xmin, xmax) y1 = - xmin*W[1]/W[2] y2 = - xmax*W[1]/W[2] plot([xmin, xmax], [y1, y2], c="k") end |
Out:draw_line (generic function with 1 method)
|
1 2 3 4 5 |
######################## # create model M = 2 # input dimension Sigma_w = 100.0 * eye(M) # prior on W |
Out:2×2 Array{Float64,2}:
100.0 0.0
0.0 100.0
|
1 2 3 4 5 6 7 8 |
######################## # create toy-data using prior model N = 50 # num of data points X = 2 * rand(M, N) - 1.0 # input values # sample observation Y Y, _ = LogisticRegression.sample_data(X, Sigma_w) |
Out:(Bool[true, false, false, false, false, true, false, false, false, false … false, false, true, true, false, false, true, true, false, false], [23.3394, 0.0910565])
|
1 2 3 4 5 6 7 |
######################## # inference alpha = 1.0e-4 # learning rate max_iter = 100000 # VI maximum iterations # learn variational parameters (mu & rho) mu, rho = LogisticRegression.VI(Y, X, M, Sigma_w, alpha, max_iter) |
Out:([8.90405, -0.76043], [3.98836, -0.616809])
|
1 2 3 4 5 |
######################## # visualize (only for M=2) visualize_surface(mu, rho, X, Y, "prediction") visualize_contour(mu, rho, X, Y) show() |